靶向铜绿假单胞菌粘肽合成酶PBP3的小分子化合物的虚拟筛选
包蓉蓉 安艳冬 黄彩敏 边榕梅 陈凯飞 宫兴文
(浙江工商大学 食品与生物工程学院,浙江杭州310012)
[摘 要] β-内酰胺类抗生素对细菌细胞壁合成有抑制作用,故可用于疾病治疗,但目前药物滥用情况严重,造成细菌较强的耐药性。细菌主要通过改变粘肽合成酶,即青霉素结合蛋白(penicillin-binding protein,PBPs)的结构和产生β-内酰胺酶等方式,对β-内酰胺类抗生素产生耐药性。在铜绿假单胞菌中,粘肽合成酶PBP3对其细胞壁的合成和菌体的存活起到至关重要的作用。研究针对铜绿假单胞菌的粘肽合成酶PBP3,通过分子对接软件DOCK6.5进行大规模虚拟筛选,以期得到具有全新结构的高亲和力的先导化合物。对含有104万个化合物的数据库进行了虚拟筛选,通过grid score进行第一轮初筛,筛选出grid score分值小于-30 kcal/mol的6万个化合物;以amber score进行第二轮筛选,筛出amber score分值小于-20 kcal/mol以下的化合物约200个;最终通过观察分析,从中挑选出4种打分高并且结构新颖的先导化合物。这四种先导化合物有望进一步开发为新型抗菌药物,用于解决铜绿假单胞菌的耐药性问题。
[关键词] 铜绿假单胞菌;粘肽合成酶PBP3;耐药性;虚拟筛选;DOCK
The Virtual Screening of Small Molecules Targeting to Penicillin-binding Protein PBP3 of P.aeruginosa
Bao Rongrong An Yandong Huang Caimin Bian Rongmei Chen Kaifeigong Xingwen
(College of Food Science and Biotechnology,Zhejianggongshang University,Hangzhou Zhejiang 310012,China)
Abstract:β-lactam antibiotics inhibit the synthesis of bacterial cell wall,thus can be used for disease treatment.However,the drug abuse is becoming serious in recent years that cause strong drug resistance of bacteria.The drug resistance of bacteria is mainly resulted from the changes of their PBP structures and the production ofβ-lactamases.The penicillinbinding protein 3(PBP3)of P.aeruginosa is essential for its survival.In this study,to obtain leads with high affinity to PBP3 of P.aeruginosa,we performed virtual screening with UCSF dock6.5 suite.The first round of screening was conducted usinggrid score.There were approximately 60,000 small molecules,with scores lower than-30 kcal/mol,were screened out from the ZINC database containing 1,040,000 compounds.The top 600 compounds were subjected to the second round of screening.Amber score was then used for the screening.Finally,200 compounds with amber scores lower than-20 kcal/mol were selected for analysis and 4 of them were selected as lead compounds for further studies.The leads have novel structures and have potential to become new agents to fight with the drug-resistance P.aeruginosa.
Key words:P.aeruginosa;Penicillin-binding protein PBPs;Drug resistance;Virtual screening;DOCK
铜绿假单胞菌(P.aeruginosa),原称绿脓杆菌,在自然界分布广泛,对人类而言,是一种条件致病菌。长期应用激素、免疫抑制剂,进行肿瘤化疗、放射治疗等导致病人免疫功能低下,以及手术后或某些治疗操作后(气管切开、保留导尿管等)的病人易导致铜绿假单胞菌感染,故其为医院内感染的重要病原菌之一,而且,近年来该细菌的耐药情况越来越严重,急需开发新的药物用于临床治疗。
β-内酰胺类抗生素是指化学结构中具有β-内酰胺环的一大类抗生素,主要包括4类:青霉素类﹑头孢菌素类﹑非典型β-内酰胺类抗生素和β-内酰胺酶抑制剂。β-内酰胺类抗生素具有共同的抗菌作用机制,主要是通过与细菌的粘肽合成酶,即青霉素结合蛋白(penicillin-binding protein,PBPs)结合而抑制细菌细胞壁的合成,属于繁殖期杀菌药。此类抗生素具有杀菌活性强、毒性低、适应证广及临床疗效好等优点,一直以来在临床上视为抗感染的首选抗菌药物。
细菌对β-内酰胺类抗生素耐药主要有以下3种机制:阻止抗生素通过细胞外膜进入细胞内,产生β-内酰胺酶,以及作用靶位结构的改变,即PBPs的改变。必要PBPs的含量变化或缺失,或其与抗生素的亲和力降低,或细菌产生缓慢结合的PBPs,或诱导性PBPs的出现等均是细菌对β-内酰胺类抗生素产生固有耐药的主要原因。
针对细菌的抗药性,可以采取的措施包括:一是联合用药,作用于不同PBPs的β-内酰胺类抗生素在联用时可产生协同作用;二是新药开发,包括改造现有药物,保留原有的对细菌靶位的作用,增强其作用机制但避免其耐药机制;三是开发辅助药物以钝化其耐药机制;四是应用细菌基因功能学以及蛋白质组学以发现作用于新靶位的新的抗生素。
从已有的化合物,包括合成化合物和天然产物中寻找药物或者先导化合物,是药物发现的一条重要途径。虚拟筛选,即分子对接,是一种基于生物靶标大分子结构的药物设计方法,其原理是通过将化合物三维结构数据库中的分子逐一与靶标分子进行“对接”,通过不断优化小分子化合物的位置、方向以及构象,寻找小分子与靶标生物大分子作用的最佳构象,计算其与生物大分子的相互作用能,对化合物数据库中所有的分子排序,从中找出可能与靶标分子结合的分子。通过并行化的分子对接方法(又称为高通量虚拟筛选),可以在几天内完成对含数十万,甚至数百万个化合物的数据库的筛选。分子对接已成为一种与高通量筛选互为补充的快速寻找先导化合物的方法。
铜绿假单胞菌的粘肽合成酶PBP3对铜绿假单胞菌的细胞壁合成和细菌的存活起着至关重要的作用。根据抗生素与PBPs结合的原理,选择铜绿假单胞菌的粘肽合成酶PBP3作为虚拟筛选的靶点,对含有104万种小分子化合物的数据库进行筛选,希望筛选出能与PBP3特异性结合的高亲和力的先导化合物,竞争性抑制PBP3底物的结合,从而阻断铜绿假单胞菌的细胞壁合成。同时,通过计算机进行大规模虚拟筛选,有利于得到具有全新结构的先导化合物,它们的结构不同于已有的β-内酰胺类抗生素(如青霉素),从而避免被耐药细菌所产生的β-内酰胺酶降解,克服现有的β-内酰胺类抗生素(如青霉素)容易被β-内酰胺酶降解所致的细菌耐药性问题。而且,这些先导化合物还可以用作β-内酰胺类抗生素的增效剂。
1. 材料和方法
1.1 程序和数据库
DOCK程序是开发最早也是目前应用最广泛的一个分子自动对接及数据库搜寻算法。该程序由美国加利福尼亚大学Kuntz等开发,选用版本为DOCK6.5。Chimera程序由加州大学旧金山分校计算机图形实验室下载(http://www.cgl.ucsf.edu/chimera)。ZINC是市售化合物中免费用于虚拟筛选的数据库。本研究的目的是开发新型抗菌药物,因此选用的是ZINC数据库中的drug-like子数据库。
1.2 准备受体和配体文件
从protein data bank网站(http://www.pdb.org/pdb/home/home.do)下载铜绿假单胞菌粘肽合成酶PBP3与头孢他啶的复合物晶体结构的pdb文件(3pbo.pdb)。然后,在Chimera中选择和删除配体,然后进行受体准备工作,包括去除溶剂、修正错误命名的氨基酸残基、加氢和加电荷等内容。在Chimera中删除复合物中除了配体以外的所有结构,保留配体。然后给配体加氢和合适的电荷,以获得配体文件。
1.3 生成球集
球集是在受体分子表面的内陷位置所产生的负像(或阴影),用于代表配体分子可能结合的位点。为保证对接的精确性,以头孢他啶的结合位点为参照,选择距离该结合位点一定半径内的球集,通过Chimera软件观察选择出来的球集,对该球集进行编辑,去除位置不佳的球体,最终得到的球集文件用作虚拟筛选时的活性位点。然后在活性位点外面生成一个盒子,用于对筛选时小分子化合物的结构和对接时的位点变化情况进行约束,以便节省虚拟筛选的运算时间。
1.4 生成grid文件
实验采用基于网格(grid)的能量打分功能。DOCK的能量打分是一种力场评分。其中,除了需要给受体文件、盒子文件和力场文件指明所在的路径以外,其余参数均用默认值。程序会分别产生以.cnt,.nrg及.bmp为扩展名的接触、能量以及碰撞计算独立文件。在实验中,网格文件命名时以“grid”为前缀,分别为grid.nrg和grid.bmp。
1.5 第一轮筛选:配体的柔性对接
第一轮筛选采用柔性对接,在柔性对接时,配体是柔性的,配体的结构是会重排的,以便与受体更好地结合。
对接时,采用grid score来评价筛选出的化合物与受体的亲和力大小。计算结束后,生成的结果文件按打分情况由高到低进行排列,每个配体分子都会给出最优的构象和结合位点信息,用Chimera等视图软件对配体的构象在受体结合位点的结合情况进行观察分析。挑选出打分高的配体分子,进入第二轮筛选。
1.6 第二轮筛选:采用amber score
Amber score可以在削减计算成本的同时仍然保持精度,配体结合部位的原子距离可以保持固定。Amber score的主要优点是在执行中,无论是配体还是受体的活性部位都可以是柔性的,允许小的结构重组,重现所谓的“诱导契合”。
由于能量优化和MD模拟很耗费时间,所以采用默认的100次能量优化和3000步MD。同时,设定距离配体5![]() 的受体氨基酸残基是柔性的,构象可以变化,以便与配体更好地契合,而其余的参数多采用默认值。
的受体氨基酸残基是柔性的,构象可以变化,以便与配体更好地契合,而其余的参数多采用默认值。
2. 结 果
2.1 受体和配体文件的准备
从protein data bank网站下载铜绿假单胞菌粘肽合成酶PBP3与头孢他啶的复合物晶体结构文件3pbo.pdb,可视化后得图4-1(a)。对受体蛋白PBP3进行去除溶剂、修正错误命名的氨基酸残基、加氢和加电荷等处理,得图4-1(b)。再对配体头孢他啶进行加氢加电荷处理,以获得配体文件,得图4-1(c)。(https://www.daowen.com)
2.2 生成球集
运行sphgen程序,产生球集,选择与距离头孢他啶10![]() 的球集。然后,观察选择出来的球集,去除位置不佳的球体,最终得到由14个球体所组成的球集,用作虚拟筛选时候的活性位点,见图4-1(d)。围绕活性位点的盒子的距离设为5
的球集。然后,观察选择出来的球集,去除位置不佳的球体,最终得到由14个球体所组成的球集,用作虚拟筛选时候的活性位点,见图4-1(d)。围绕活性位点的盒子的距离设为5![]() ,以保障盒子的大小足够容纳小分子配体而且不影响计算速度见图4-1(d)。
,以保障盒子的大小足够容纳小分子配体而且不影响计算速度见图4-1(d)。
2.3 第一轮虚拟筛选:grid score
对含104万个化合物的ZINC数据库进行了虚拟筛选,第一轮筛选采用grid score来评价筛选出的化合物与受体的亲和力大小。最终,得到打分在-30 kcal/mol以上的化合物约6万个,从中挑选出打分高的600个化合物进入第二轮筛选。
2.4 第二轮筛选:amber score
第二轮筛选用amber score。Amber score可以在削减计算成本的同时仍然保持精度,主要优点是在执行中,无论是配体还是受体的活性部位都可以是柔性的,允许小的结构重组,重现所谓的“诱导契合”。经过运算,最终筛出打分高于-20 kcal/mol的化合物约200个,选取4种打分理想的化合物进行分析(见表4-1)。
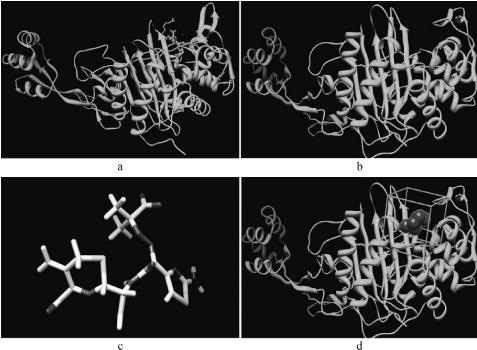
图4-1 生成受体、配体和球集文件
表4-1 筛选出的4种化合物的打分情况
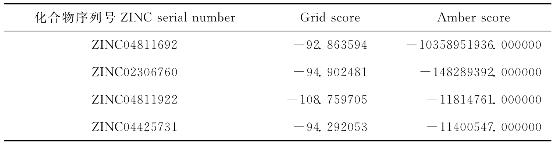
从表4-1中可以看到,在第一轮筛选中,ZINC04811922的grid score是最高的,但amber score却不是最高的;而在第二轮筛选中,ZINC04811692的amber score是最高的,但第一轮的grid score却不是最高的。这是由于第二轮的amber score才是精筛,因为在amber score筛选时受体和配体的相互作用部分都是柔性的,这样才能得到更精确的结果。
对这4种化合物与受体结合后的情况进行观察分析,可以看出,受体的活性位点呈口袋形状,配体化合物结合在口袋部位,而且形态匹配较好(图4-2)。

图4-2 先导化合物与受体的结合情况
另外,对以上4种化合物与受体之间的相互作用情况进行了分析,可以看到这四种化合物可以与周围的氨基酸残基形成较多的疏水作用和氢键作用(图4-3),这可能是这四种化合物与受体具有较高亲和力的原因。由图4-3中可以发现,ZINC04811692与PBP3有一个疏水作用和2个氢键,ZINC02306760与PBP3有5个疏水作用,ZINC04811922与PBP3有3个疏水作用和3个氢键,ZINC04425731与PBP3有4个疏水作用和3个氢键。虽然ZINC04811692与PBP3的相互作用最少,但amber打分却最高,这可能与这些相互作用力的距离有关。此外,虚拟筛选进行的是理论计算,而最终的亲和力大小,还要通过具体的试验测定来确定。由于小分子化合物与大分子蛋白质之间的亲和力测定需要表面等离子共振(SPR)等高端仪器来完成,并且耗时较长。因此,这部分内容未包含在本研究范围内,将在后续的研究中完成。
通过查询CA和ZINC数据库,未发现这四种化合物的相关研究报道,说明是新的化合物。同时,这4种化合物的结构与已有的β-内酰胺类抗生素不同,因此,可以避免被耐药细菌所产生的β-内酰胺酶降解,克服现有的β-内酰胺类抗生素(如青霉素)容易被β-内酰胺酶降解所致的细菌耐药性问题。而且,这些先导化合物还可以用于抑制耐药细菌所产生的β-内酰胺酶,用作β-内酰胺类抗生素的增效剂。因此,这4种先导化合物有望进一步开发为新型抗菌药物,用于解决细菌的耐药性问题。
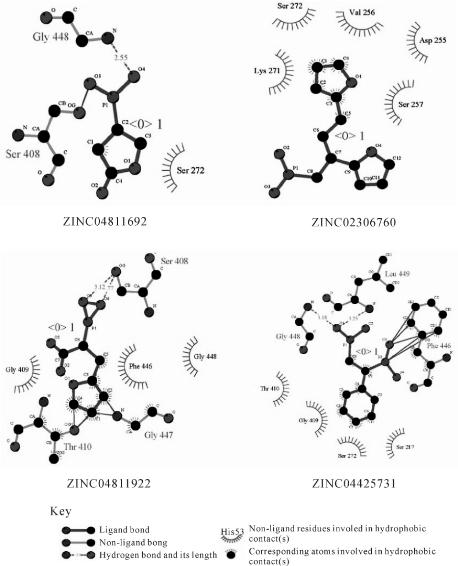
图4-3 先导化合物与受体蛋白之间的相互作用分析
3. 讨 论
计算机辅助药物设计是新药开发的一个重要组成部分,其中,基于受体结构的分子对接由于适用于大规模数据库筛选而被广泛应用。通过计算机进行大规模虚拟筛选,利用计算机强大的计算能力从已建立的大规模化合物的三维数据库(含上百万个化合物)中搜寻与靶标生物大分子活性部位或者结合部位相匹配的化合物,其目的是快速发现有苗头的化合物,集中目标进行攻关。通过计算机进行虚拟筛选,可以节省大量的人力、物力和财力,加强药物研发进度,并且有利于得到具有全新结构的先导化合物。
在本次研究中,第一轮筛选采用了grid score,进行grid score筛选时,配体是柔性的,受体是刚性的,从而保证了筛选的速度而且也可以筛选到形态匹配较好的配体分子。第二轮筛选用amber score,amber score可以在削减计算成本的同时仍然保持精度,优点是在执行中,无论是配体还是受体的活性部位都可以是柔性的,允许小的结构重组,重现所谓的“诱导契合”。最后,通过对筛选出的化合物进行进一步分析,可以得到亲和力高、结构新颖的先导化合物,用于后续研究。
本研究根据β-内酰胺类抗生素的作用机理,靶向铜绿假单胞菌的粘肽合成酶PBP3,对含有104万种小分子化合物的数据库进行虚拟筛选,目标是筛出能与PBP3特异性结合的高亲和力的先导化合物,通过竞争性抑制PBP3底物的结合从而阻断铜绿假单胞菌的细胞壁合成。全新的结构可以避免被耐药细菌所产生的β-内酰胺酶降解,克服现有的β-内酰胺类抗生素(如青霉素)容易被β-内酰胺酶降解所致的细菌耐药性问题,而且,这些先导化合物还可以用作β-内酰胺类抗生素的增效剂。
对含有104万个化合物的数据库中进行了虚拟筛选,通过grid score进行第一轮初筛,得到grid score分值小于-30 kcal/mol化合物约6万个,接着以amber score进行第二轮筛选,筛出amber score分值小于-20 kcal/mol的化合物约200个。通过进一步的观察分析,最终挑选出4种亲和力高并且结构新颖的先导化合物。这4种先导化合物有望进一步开发为新型抗菌药物,用于解决铜绿假单胞菌的耐药性问题。
[参考文献]
[1]莫岚,王其南,审棱.青霉素结合蛋白与细菌对β-内酰胺类抗生素耐药机制的研究进展[J].国外医药抗生素分册,1996,17(3):187-191.
[2]武正兰.青霉素作用机制和不合理应用分析[J]医药导报,2001,20(4):259-260.
[3]Nancock R E.Resistence mechanism in pseudomonas aeruginosa and other nonfermentativegram-negative bacterial[J].Clin infect Dis,2001,27(suppl):93-95.
[4]张城,李伟章,恽榴红.用组合化学建立天然产物类似物库[J].化学进展,2003,15 (3):194-203.
[5]Good A C,Krystek S R,Mason J S.High-throughput and virtual screening-core lead discovery technologies move towards integration[J].Drug Diseov Today,2000,5(12):61-69.
[6]朱伟,陈可冀,徐筱杰.计算机药物虚拟筛选技术在中医药领域中的应用前景[J].中国中西医结合杂志,2007,27(3):263-266.
[7]罗小民,蒋华良,沈建华,等,药物分子设计研究进展[J].中国科学院院刊,2003(4):255-259.
[8]徐为人,汤立达,符海霞.分子模拟与新药虚拟评价[J].中国药理通讯,2009,26(2):76-76.
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。