实验目的
1.理解酵母双杂的原理。
2.掌握酵母双杂的实验方法。
实验原理
酵母双杂交系统(Yeast-two-hybrid system)利用了真核生物转录调控因子的组件式结构特征,这些蛋白质往往由两个或两个以上相互独立的结构域构成,其中DNA结合结构域(DNA binding domain,BD)和转录激活结构域(activation domain,AD)是转录激活因子发挥功能所必需的。单独的BD能与特定基因的启动区结合,但不能激活基因的转录,而由不同转录调控因子的BD和AD所形成的杂合蛋白却能行使转录的功能。实验中,首先运用基因重组技术把已知蛋白的DNA序列连接到带有酵母转录调控因子(常为GAL1、GAL4或GCN1)DNA结合结构域编码区(BD)的表达载体上。导入酵母细胞中使之表达带有DNA结合结构域的杂合蛋白,与报告基因上游的启动调控区相结合,准备作为“诱饵”捕获与已知蛋白相互作用的基因产物。此时,若将已知编码转录激活结构域(AD)的DNA分别与待筛选的cDNA文库中不同插入片段相链接,获得“猎物”载体,转化含有“诱饵”的酵母细胞。一旦酵母细胞中表达的“诱饵”蛋白与“猎物”载体中表达的某个蛋白质发生相互作用,不同转录调控因子的AD和BD结构域就会被牵引靠拢,激活报告基因的表达。
实验用品
1.材料:拟南芥AtAP3与AtPI的开放阅读框。
2.器材:培养箱,摇床,PCR仪,电泳仪,电泳槽,凝胶成像系统,超净工作台。三角瓶,离心管,枪头,烧杯,三角瓶,培养皿,镊子,剪刀,酒精灯。
3.试剂:酵母菌株AH109,pGBKT7载体(Binding domain)(Trp),pGADT7载体(Activation domain)(Leu);20 mg/mL X-gal;Nde I,EcoR I和BamH I限制性内切酶;各种SD培养基;Z Buffer,ONPG,YPAD培养基;KAc缓冲液(pH 4.8);溶液Ⅰ,溶液Ⅱ,溶液Ⅲ;PEG(50%);LiAC(1 mol/L);ssDNA(10 mg/mL)。
(1)Z Buffer的配方:
Na2HPO4 8.5g
NaH2PO4·H2O 5.5 g
KCl 0.75 g
MgCl2·7H2O 0.246 g
补ddH2O 1 L
(2)YPAD培养基的配方:
酵母提取液 10 g
消化蛋白质 20 g
NaOH 0.1 g
补ddH2O 900 mL
高温灭菌后补加10 mL灭过菌的20%(W/V)葡萄糖。
(3)溶液Ⅰ的配方:
葡萄糖 50 mmol/L
Tris-HCl(pH 8.0) 25 mmol/L
EDTA(pH 8.0) 10 mmol/L
(4)溶液Ⅱ的配方:
NaOH 0.2 mol/L
SDS 1%(W/V)
(5)溶液Ⅲ的配方:
KOAc(5 mol/L) 60 mL
冰醋酸 11.5 mL
ddH2O 28.5 mL
(6)10×SD溶液:
L-尿嘧啶(uracil) 200 mg/L
L-腺嘌呤亚硫酸盐(Adenine hemisulfate salt) 200 mg/L
L-盐酸精氨酸(Arginine HCl) 500 mg/L
L-天冬氨酸(Aspartic acid) 1000 mg/L
L-谷氨酸(Glutamic acid) 1000 mg/L
L-异亮氨酸(Isoleucine) 300 mg/L
L-亮氨酸(Leucine) 1000 mg/L
L-盐酸赖胺酸(Lysine HCl) 500 mg/L
L-蛋氨酸(Methionine) 200 mg/L
L-苯丙氨酸(Phenylalanine) 500 mg/L
L-丝氨酸(Serine) 400 mg/L
L-苏氨酸(Threonine) 2000 mg/L
L-酪氨酸(Tyrosine) 500 mg/L
L-缬氨酸(Valine) 1500 mg/L
L-色氨酸(Tryptophan) 500 mg/L
L-组氨酸(Histidine HCl monohydrate) 200 mg/L
配置营养缺陷培养基时不能加相应的氨基酸。灭菌后置于4℃可保存1年。
(7)酵母筛选用营养缺陷培养基SD/-Trp(pH 5.8):
酵母氮源(不含氨基酸) 6.7 g
10×SD(不含有-Trp) 100 mL
NaOH 0.1 g
琼脂 20 g
dH2O 至900 mL
高温灭菌后补加100 mL灭过菌的20%(W/V)葡萄糖。
(8)酵母筛选用营养缺陷培养基SD/-Leu(pH 5.8):
酵母氮源(不含氨基酸) 6.7 g
10×SD(不含有-Leu) 100 mL
NaOH 0.1 g
琼脂 20 g
dH2O 至900 mL
高温灭菌后补加100 mL灭过菌的20%(W/V)葡萄糖。
(9)酵母筛选用2营养缺陷培养基SD/-Trp-His(pH 5.8):
酵母氮源(不含氨基酸) 6.7 g
10×SD(不含有-Trp-His) 100 mL
NaOH 0.1 g
琼脂 20 g
dH2O 至900 mL
高温灭菌后补加100 mL灭过菌的20%(W/V)葡萄糖。
(10)酵母筛选用3营养缺陷培养基SD/-Leu-Trp-His(pH 5.8):
酵母氮源(不含氨基酸) 6.7 g
10×SD(不含有-Leu-Trp-His)100 mL
NaOH 0.1 g
琼脂 20 g
dH2O 至900 mL
高温灭菌后补加100 mL灭过菌的20%(W/V)葡萄糖。
实验程序
1.AtAP3与AtPI的克隆
在NCBI中查找拟南芥AtAP3与AtPI的全长序列,PCR扩增出AtAP3与AtPI的完整读码框。根据载体pGADT7和pGBKT7载体的多克隆位点(图43-1),在AtAP3片段的5’端与3’端引入相应的酶切位点;在AtPI片段的5’端与3’端引入相应的酶切位点。
AtAP3 PCR的反应体系(50 μL):
0.5 μL ExTaq DNA聚合酶(Takara)
5 μL 10×Ex buffer
1 μL dNTP(10 mmol/L)
1 μL F Primer(10 pmol/L)(如Nde I)
1 μL R Primer(10 pmol/L)(如EcoR I)
1 μL cDNA
40.5 μL ddH2O
AtPI的反应体系(50 μL):
0.5 μL ExTaq DNA聚合酶(Takara)
5 μL 10×Ex buffer
1 μL dNTP(10 mmol/L)
1 μL F Primer(10 pmol/L)(如Nde I)
1 μL R Primer(10 pmol/L)(如BamH I)
1 μL cDNA
40.5 μL ddH2O
PCR反应程序:
Step 1 94 °C 3 min
Step 2 94 °C 30 s
Step 3 56 °C 30 s
Step 4 72 °C 1 min
From step 2 to step 4×35 cycles
Step 5 72 °C 10 min(www.daowen.com)
取PCR产物4 μL进行电泳检测。TianGEN PCR回收试剂盒纯化PCR产物。
2.ORF片段和载体质粒双酶切反应
AtAP3反应体系(50 μL):
5 μL 10×H buffer
2.5 μL Nde I
2.5 μL EcoR I
8 μL cDNA
32 μL ddH2O
37 °C,过夜酶切。TianGEN PCR产物纯化试剂盒纯化酶切产物。载体酶切反应同上。
AtPI反应体系(50 μL):
5 μL 10×K buffer
2.5 μL Nde I
2.5 μL BamH I
8 μL cDNA
32 μL ddH2O
37 °C,过夜酶切。TianGEN PCR产物纯化试剂盒纯化酶切产物。载体酶切反应同上。
3.ORF片段和载体质粒链接反应
反应体系(10 μL):
1 μL 10×T4 ligation buffer
1 μL T4 DNA ligase(Takara)
6 μL insert DNA
2 μL plasmid DNA
16 °C,过夜链接。
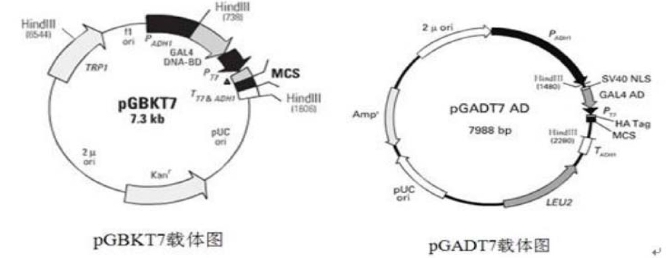
图43-1 pGBKT7与pGADT7载体图
4.载体构建阳性克隆的筛选与鉴定
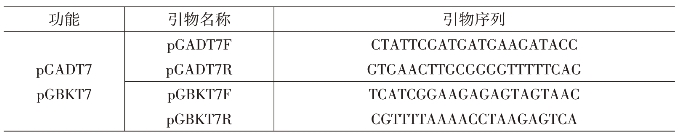
链接产物的转化、克隆、筛选与鉴定方法同实验四十。pGADT7链接产物的筛选用的抗生素为100 μg/mL Amp,pGBKT7筛选用的抗生素为50 μg/mL Kan。阳性克隆鉴定引物为载体引物pGADT7F、pGADT7R和pGBKT7F、pGBKT7R。所得阳性克隆由华大基因测序验证。验证准确的克隆,提取质粒备用。引物序列信见表43-1。
表43-1 实验中所需的引物序列
5.载体质粒的提取
SDS碱裂解法——质粒提取。
(1)将菌液倒入2 mL离心管,4 °C,12 000 r/min,离心1 min,收集菌体。
(2)加入预冷的100 μL溶液I,涡旋沉淀,使菌体混匀。
(3)将离心管置于冰上,加入200 μL溶液Ⅱ(新配制的),轻轻颠倒几次,混匀,冰浴5 min,切勿震荡。
(4)加入冰冷的150 μL溶液Ⅲ,反复颠倒数次。冰浴10 min以上。
(5)加入等体积的氯仿∶异丙醇=24∶1混匀后,4 °C,12 000 r/min,离心10 min。
(6)将上清转移至另一个离心管中,重复步骤5。
(7)将上清转移至另一个离心管中,加入2倍体积的无水乙醇。-20 °C沉淀1 h。
(8)4 °C,12 000 r/min,离心20 min,沉淀即为质粒。
(9)75%乙醇洗沉淀2次,每次5 min,残液吸净后吹干。
(10)溶于合适体积的无菌去离子水中,-20 °C保存。
6.酵母感受态细胞制备
(1)30 °C,在YPAD平板上划线培养酵母菌种AH109,2~3 d。
(2)挑选单克隆于5 mL YPAD液体培养基中,30 °C,200 r/min,过夜培养。
(3)取2 mL过夜培养物加入100 mL YPAD液体培养基中,30 °C,200 r/min,培养至OD值为0.4~0.6。
(4)转入50 mL离心管中,室温,4000 r/min,离心5 min,收菌。
(5)用25 mL ddH2O重悬菌体,室温,4000 r/min,离心5 min,收菌。25 mL ddH2O再清洗一次。
(6)用1 mL LiAC(100 mmol/L)重悬菌体,转入1.5 mL离心管中。
(7)快速离心10 s,0.5 mL LiAC(100 mmol/L)重悬菌体。分装至1.5 mL离心管中(50 μL/管),置冰上备用。
7.LiAC介导的小规模酵母转化
(1)在酵母感受态细胞中加入如下试剂:
240 μL PEG(50%)
36 μL LiAC(1 mol/L)
5 μL ssDNA(10 mg/mL)(沸水变性10 min,立即冰浴)
70 μL ddH2O。将溶液混匀。
(2)向上述细胞中加入AD和BD质粒各2 μL,充分混匀。
(3)将上述混合物30 °C温浴30 min后,加入35 μL DMSO。轻轻颠倒混匀。
(4)42 °C热激20~25 min后,立即冰浴5 min。
(5)12 000 r/min,离心1min,收菌。
(6)用1000 μL的ddH2O清洗1次。
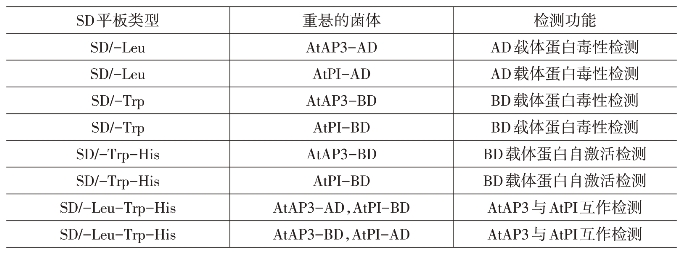
(7)用150 μL ddH2O重悬菌体,涂于相应的SD平板(见表43-2)。
(8)30 °C,倒置培养2~7 d。
表43-2 SD平板类型与对应的重悬菌体
8.蛋白互作检测
(1)AD载体蛋白毒性检测:SD/-Leu。
分别将AtAP3-AD与AtPI-AD菌体涂于SD/-Leu的平板上,如果长出白色菌体克隆,说明AtAP3-AD与AtPI-AD蛋白无自毒性,可进一步用于蛋白互作检测。
(2)BD载体蛋白毒性检测:SD/-Trp。
分别将AtAP3-BD与AtPI-BD菌体涂于SD/-Trp的平板上,如果长出白色菌体克隆,说明AtAP3-BD与AtPI-BD蛋白无自毒性,可进一步用于蛋白互作检测。
(3)BD载体蛋白自激活检测:SD/-Trp-His。
分别将AtAP3-BD与AtPI-BD菌体涂于SD/-Trp-His的平板上,如果没有长出白色的菌体克隆,说明AtAP3-BD与AtPI-BD蛋白无自激活,可进一步用于蛋白互作检测。
(4)互作检测:SD/-Leu-Trp-His。
将AtAP3-AD、AtPI-BD菌体和AtAP3-BD、AtPI-AD菌体涂于SD/-Leu-Trp-His平板上,如果长出白色菌体克隆,说明AtAP3与AtPI有互作。进一步加入2 mL X-gal,30℃培养过夜,如有变蓝反应,可进一步说明AtAP3与AtPI两转录因子有相互作用。
9.基于分光光度法的双酵母β-galactosidase活性分析
用双酵母β-galactosidase活性分析的方法能检验转录因子间蛋白互作的强弱。
(1)实验当天用Z Buffer溶解(含27 μL β-巯基乙醇)ONPG并振荡1~2 h,浓度为4 mg/mL。
(2)准备选择性SD培养基中过夜培养的菌液5 mL,并涡旋0.5~1 min。
(3)将2 mL加入8 mL YPAD培养基中。30℃,250 r/min,揺菌培养3~5 h至细胞对
数期(OD600=0.5~0.8)。
(4)每个样准备3个重复,即3个1.5 mL EP管,每管加入1.5 mL菌液,14 000 r/min离心30 s。
(5)除去上清,每管加入1.5 mL Z Buffer并涡旋至完全悬浮。
(6)收集细胞除上清,重悬于300 μL Z Buffer(此时浓缩比例为5倍)。
(7)吸取100 μL于一新鲜的EP管,置液氮中反复冻融2次,直至细胞破碎。
(8)设一个对照只加入100 μL Z Buffer。
(9)往每个反应管和对照管加入0.7 mL Z Buffer+ß-mercaptoethanol(巯基乙醇)。
(10)每个管中马上加入160 μL ONPG,30℃温浴,并开始计时。
(11)液体变为黄色后,记录所用的时间,并加入0.4 mL,1 mol/L Na2CO3终止反应。
(12)14 000 r/min离心10 min,取上清于干净的试管中。
(13)用对照在420 nm校准分光光度计,检测每个样品的OD420。
(14)最后计算β-galactosidase值。β-galactosidase units=1000×OD420/t×V×OD600)。t=反应所用的时间(min),V=0.1 mL×浓缩因子。
实验注意事项
1.蛋白互作检测时,需要有AD载体蛋白毒性检测与BD载体蛋白毒性检测,以及BD载体蛋白的自激活检测。
2.载体与基因在链接前注意限制性内切酶的选择。
思考题与作业
1.蛋白互作检测时,为什么需要有AD载体蛋白毒性检测与BD载体蛋白毒性检测?
2.双酵母β-galactosidase活性分析有什么作用?
Zhang S H,Zhang J S,Zhao J,et al.Distinct subfunctionalization and neofunctionalization of the Bclass MADS-box genes in Physalis floridana[J].Planta,2015,241:387–402.
Gong PC,Li J,He CY.Exon junction complex(EJC)core genes play multiple developmental roles in Physalis floridana[J].Plant Molecular Biology,2018,98:545-563.
Li Q X,Huo Q D,Wang J,et al.Expression of B-class MADS-box genes in response to variations in photoperiod is associated with chasmogamous and cleistogamous flower development in Viola philippica.BMC Plant Biology,2016,6:151.
朱玉贤,李毅.现代分子生物学[M].2版.北京:高等教育出版社,2002.
(李巧峡)
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。