1.微生物菌群结构多样性
对H-D_L5、L-D_L5、H-D_H9和L-D_H9四个样品的古菌和细菌的16S rRNA基因进行了PCR扩增,并对扩增后的DNA片段进行高通量测序,分析处理所有的测序结果,去掉空载体、测序质量差的序列以及嵌合体序列后,古菌和细菌群落分别得到了2 004~5 883和1 349~34 117条高质量序列。之后,通过与RDP数据库的已知序列进行比对,对所获得的序列进行分类学分析。
图6-6显示了各DNA样品文库预测古菌和细菌基因型丰度的稀疏化分析结果。当预测基因型丰度(Observed species)随可分析序列数量(Sequences per sample)的变化进入平台期,即可认为文库容量已经足以分析其丰度[204]。可见,除L-D_H9的细菌序列分析尚未进入平台期外,其他样品序列分析均显示,样品文库的库容已经足以分析其中微生物群落的多样性。由图6-6可见,细菌的基因型丰度要远高于古菌,说明细菌群落结构比古菌更为复杂。而标记DNA中细菌和古菌的基因型丰度高于未标记DNA,说明整个微生物群落中仅小部分成员处于代谢活跃状态。低氨浓度下标记DNA中细菌和古菌的基因型丰度高于高氨浓度下,说明氨胁迫降低了功能微生物种群结构的多样性,仅耐氨胁迫的微生物种群处于代谢活跃状态。
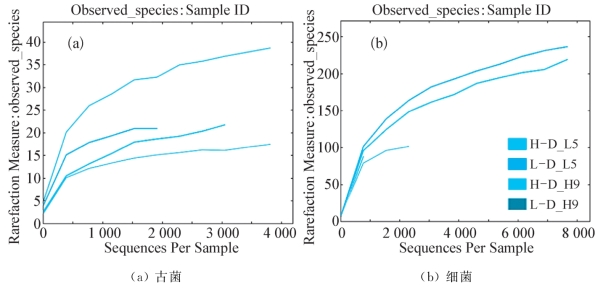
图6-6 预测古菌和细菌基因型丰度的稀疏曲线
如表6-3所示,不同DNA样品中微生物种群结构的多样性特征也可以通过香农多样性指数(H)得到验证。
表6-3 各DNA组分中微生物菌群结构的香农多样性指数H
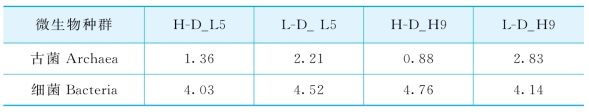
注:“L-D_L5”和“H-D_L5”分别代表:TAN 19 mmol/L下13C标记实验培养5 d后,累计产甲烷量达到75%时的轻DNA组分和重DNA组分;L-D_H9和H-D_H9分别代表:TAN 500 mmol/L下13C标记实验培养9 d后,累计产甲烷量达到50%时的轻DNA组分和重DNA组分。
2.古菌域(Domain Archaea)
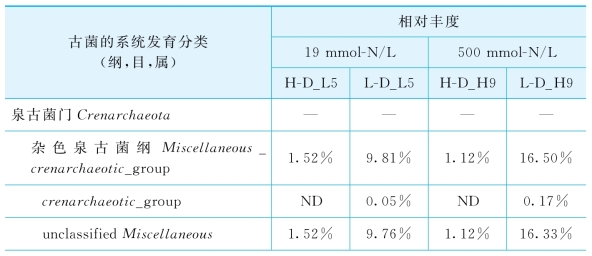
各DNA样品中,古菌的基因型在纲、目和属分类水平的分布见表6-4。
表6-4 古菌的基因型在纲、目和属分类水平的分布
 (https://www.daowen.com)
(https://www.daowen.com)
续表
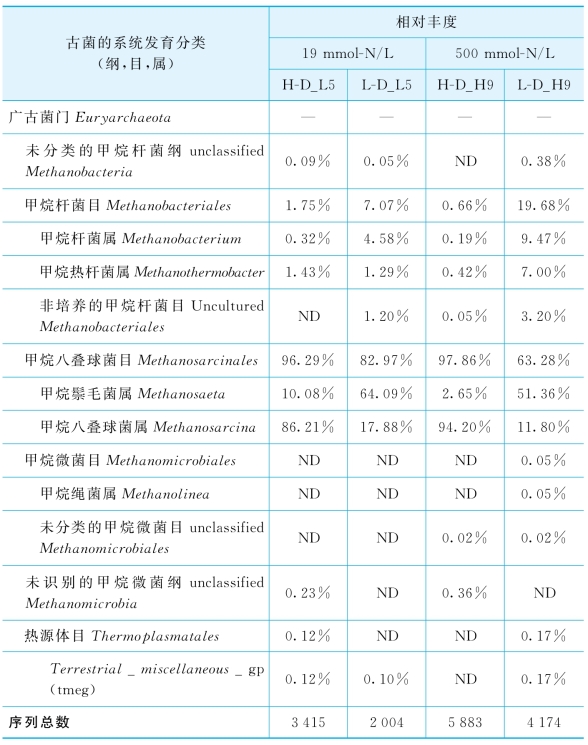
重DNA组分的古菌基因文库中,数量最多的为甲烷八叠球菌属Methanosarcina,其序列数目分别在H-D_L5和H-D_H9文库中占序列总数的86%和95%;甲烷鬃毛菌属Methanosaeta的丰度居于第二位,分别占序列总数的10%和3%。而在轻DNA基因文库中,甲烷鬃毛菌属Methanosaeta的丰度最高。甲烷杆菌目的成员,如甲烷杆菌属Methanobacterium和甲烷热杆菌属Methanothermobacter,仅在轻DNA基因文库中少量存在;同样,甲烷微菌目Methanomicrobiales仅在L-D_H9文库中被少量检出(0.05%)。由此可见,甲烷八叠球菌属为乙酸快速降解阶段的优势菌群,且具有较高的氨胁迫耐受性;甲烷鬃毛菌属在原微生物体系内具有极高的丰度,但在高浓度乙酸的降解过程中并不活跃;氢营养型产甲烷菌,包括甲烷杆菌目和甲烷微菌目,未在乙酸的厌氧转化中起到显著作用。
隶属于泉古菌门Crenarchaeota的序列分别占轻DNA基因文库的9.8%~16.5%和重DNA基因文库的1.2%~1.5%。有文献报道,泉古菌门的一些成员具有氧化氨的功能[205]。然而,在本实验的整个培养过程中,未观测到TAN的显著下降。因此,泉古菌在此厌氧消化体系中的功能尚不明确。
3.细菌域(Domain Bacteria)
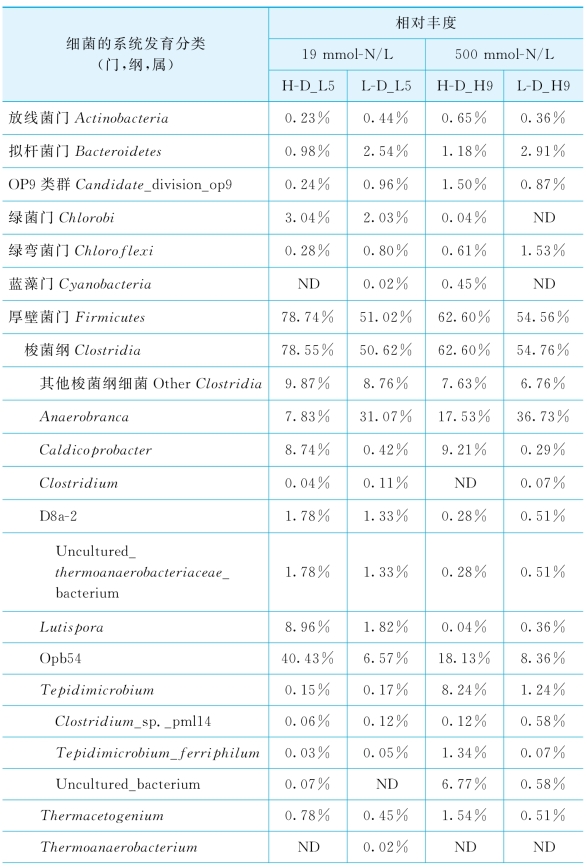
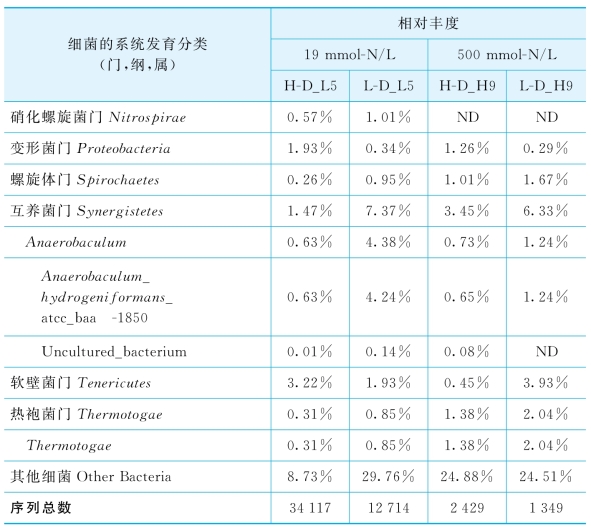
由表6-5可知,在整个微生物群落结构中,无论是低氨浓度还是高氨浓度下,梭菌纲Clostridia均占据了绝对优势,其序列数占总序列的51%~79%;在属于梭菌纲的序列中,Opb54是重DNA基因文库中丰度最高的细菌类群,在低氨浓度和高氨浓度培养下分别占40%和18%;另一个主要分布在重DNA中的细菌种群为Caldicoprobacter属,约占对应文库总序列数的9%;而低丰度的变形菌门Proteobacteria则仅存在于重DNA的基因文库中。上述细菌种群在高氨浓度和低氨浓度反应器中获得的重DNA基因文库中均有存在,表明这些细菌种群可能参与了13C的代谢,但对氨胁迫不敏感。
分别占总序列数8%和0.5%~1.5%的Tepidimicrobium属和Thermacetogenium属的细菌种群,大部分存在于高氨浓度反应器的重DNA基因文库中,表明这些细菌在氨胁迫下具有对乙酸的竞争优势;而被归类为Lutispora属的细菌种群,则大部分存在于低氨浓度反应器的重DNA基因文库,表明其代谢活力易受氨胁迫的影响;Anaerobranca属的细菌种群则在轻DNA基因文库中丰度最高,该细菌种群可能与污泥的内源呼吸有关。
表6-5 细菌的基因型在纲、目和属分类水平的分布
续表
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。






