1.qPCR反应体系
为监测关键微生物种群的动态变化,将基于16S rRNA基因设计的引物作为生物标记物,采用Taqman qPCR技术定量分析不同微生物类群,包括氢营养型的甲烷杆菌目Methanobacteriales(MBT-set)和甲烷微菌目Methanomicrobiales(MMB-set),严格乙酸营养型的甲烷鬃毛菌科Methanosaetaceae(Mst-set)和多营养型的甲烷八叠球菌科Methanosarcinaceae(Msc-set),以及总细菌Bacteria(BAC-set)和古菌Archaea(ARC-set)。其中,还采用SYBR Green方法检测了甲烷囊菌属(Methanculleus-set)和甲烷八叠球菌属(Methanosarcina-set)的数量。另外,将基于功能基因设计的引物FTHFS-set和ACS-set作为生物标记物,定量化分析了共生乙酸氧化细菌的丰度。qPCR中所使用的引物和探针信息见表2-6。
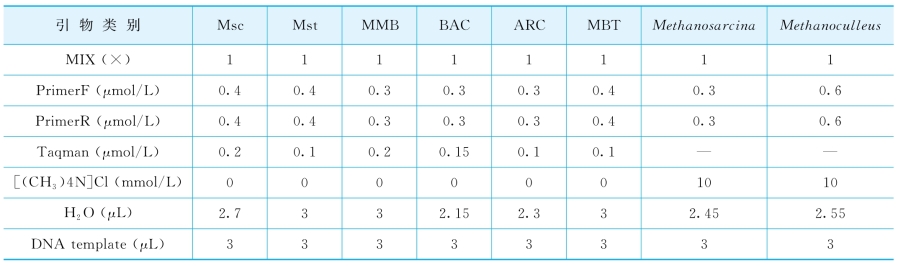
Taqman方法中使用了IQTMSupermix试剂盒(BioRad,U.S.A.),而SYBR Green方法中使用了IQTMSYBR®Green Supermix试剂盒(BioRad,U.S.A.)作为反应缓冲液(其中已包含PCR所需酶和各种离子)。反应体积为15μL,Taqman qPCR反应体系由缓冲液、引物和探针、DNA模板和水组成,SYBR Green qPCR反应体系由缓冲液、引物、DNA模板、[(CH3)4N]Cl溶液和水组成。qPCR反应体系的构成见表2-7。
反应在CFX96 TouchTM型实时定量PCR仪(Bio-Rad,Hercules,California,U.S.A.)内进行,反应过程和结果采用软件Bio-Rad CFX Manager进行控制和分析。表2-8显示了各qCPR反应的程序。反应程序参考文献[162,168,175,190]。
2.标准曲线
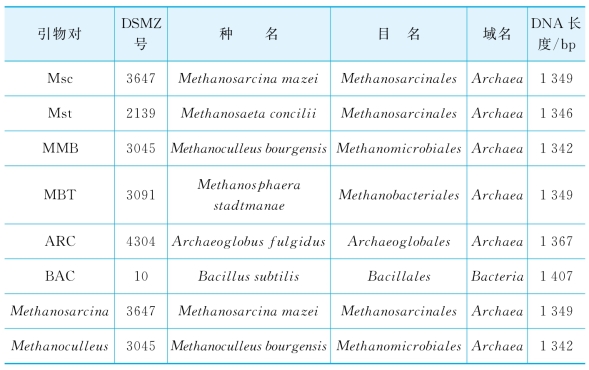
在定量化分析各产甲烷菌种群、总细菌和古菌时,采用了各自模式菌株的质粒DNA作为标准品制作标准曲线,DNA标准品信息见表2-9。
对于共生乙酸氧化细菌,由于缺少模式菌株的DNA,因此只能进行相对定量分析,制作标准曲线所用的DNA模板为样品DNA。采用FTHFS-set和ACS-set引物进行PCR扩增,反应体系为50μL,包括:1×PCR缓冲液(5μL),0.75 U的Taq DNA聚合酶(0.6μL),0.2 mmol/L核苷酸(1μL),0.40μmol/L引物(各0.5μL),1.5 mmol/L MgCl2(3μL)。PCR程序为:94℃3 min,30个循环(94℃45 s,55℃1 min,72℃2 min),72℃延伸7 min。扩增产物纯化回收后,在紫外分光光度计下测试核酸浓度,即OD值。根据式(2-2)计算产物的拷贝数[191]。然后,依次以10n进行稀释,再进行qPCR扩增,扩增的反应体系见表2-9,反应程序参照表2-8。扩增得出梯度浓度样品的CT值,据此建立标准曲线。
表2-6 qPCR 扩增中的引物序列
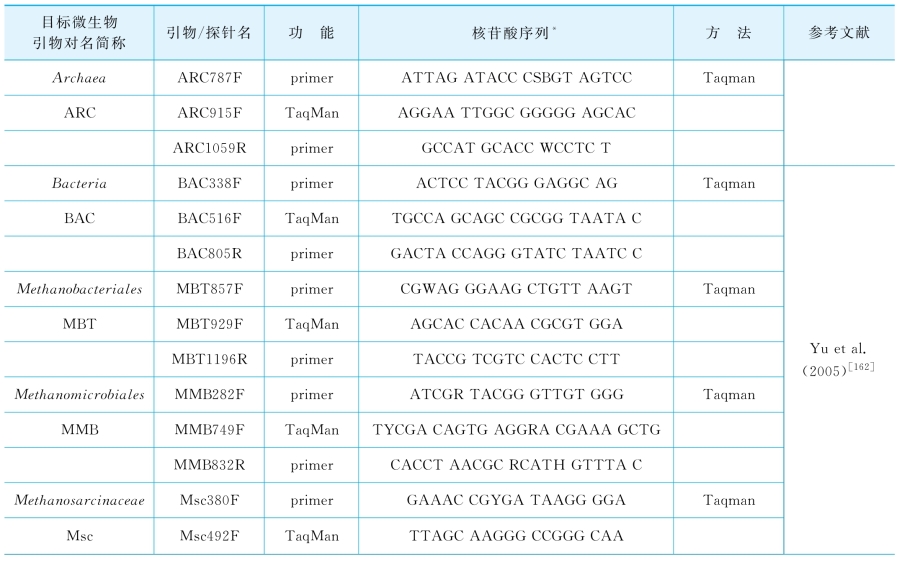
续表
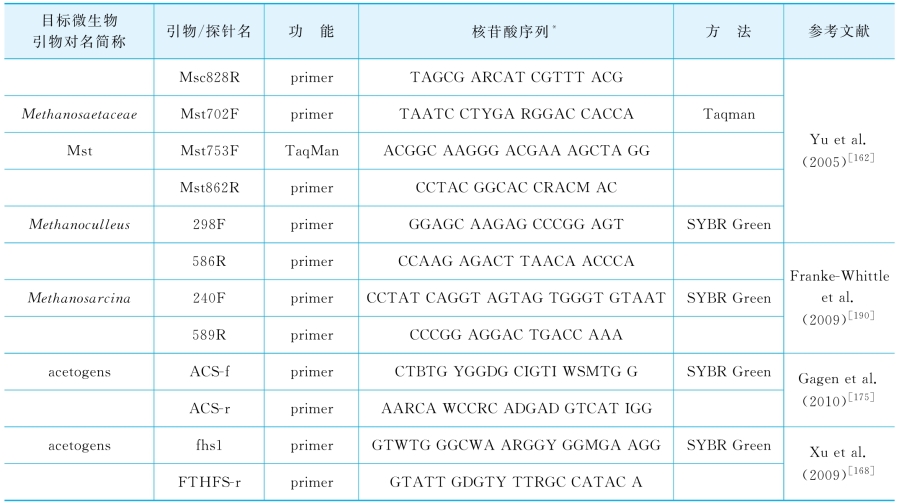
注:*核酸序列中,B代表C、G 或T;H 代表A、C 或T;M 代表A 或C;R 代表A 或G;W 代表A 或T;Y 代表T 或C。
表2-7 qPCR 反应溶液体系
 (www.daowen.com)
(www.daowen.com)
表2-8 qPCR 扩增程序[162,168,175,190]
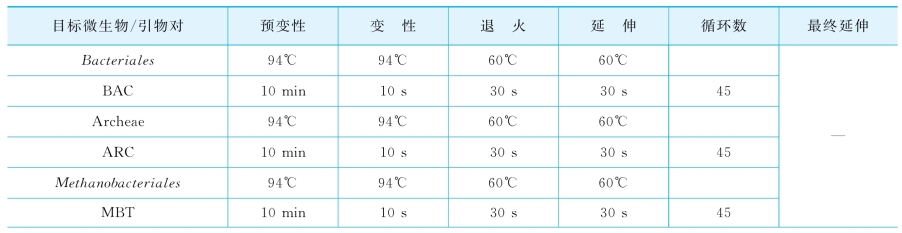
续表
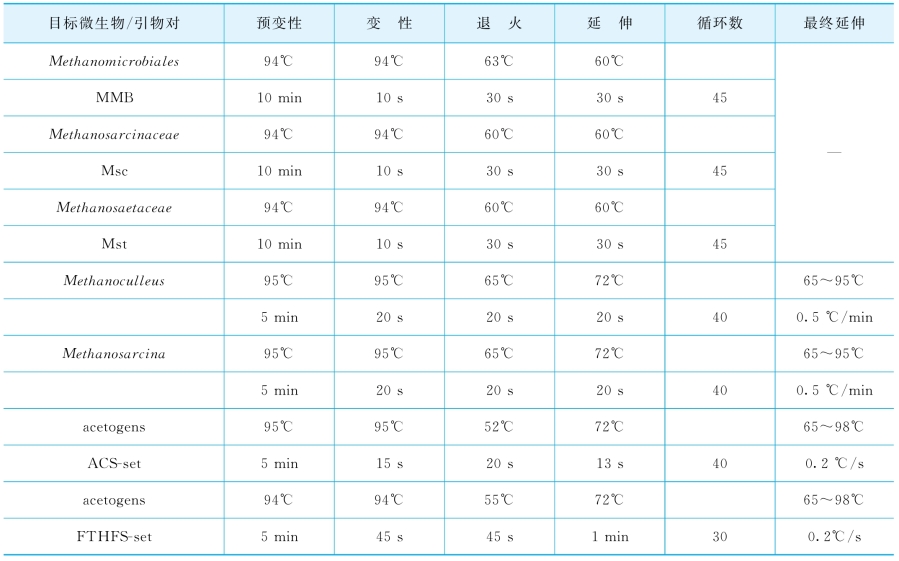
表2-9 构建qPCR标准曲线的DNA标准品信息

3.DNA纯化
酚氯仿提取法获得的DNA需进行纯化。纯化方法如下:
(1)DNA收集,采用1.0%琼脂糖凝胶电泳(10 g/L琼脂糖,1×TAE)使DNA聚集,形成目的DNA片段。
(2)将含有目的DNA片段的琼脂糖凝胶切下(尽量切除多余部分),并放入1.5 mL离心管中。
(3)采用TIANgel Midi DNA纯化试剂盒(天根生化科技有限公司,北京,中国),按照说明对DNA进行纯化。
(4)采用紫外分光光度计(NanoDrop2000c,Thermo Scientific.,U.S.A.)检测回收得到的DNA溶液的浓度。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。