随着厌氧消化工艺的推广和越来越多的工程化应用,对于厌氧产甲烷微生态的研究方法也得到了逐步发展。从最初利用常规方法对代谢过程中物质(包括底物、中间产物和最终产物)转化的监测,发展到利用多种分子生物学技术对微生物种群结构的分析,以及利用同位素示踪技术对于厌氧甲烷化代谢途径的监测,可实现对不同情景下产甲烷微生态的演变规律进行多角度全方位的探索。分子生物学技术和同位素标记示踪方法的快速发展,使我们对生态系统的研究摆脱了完全黑箱模式,对于厌氧消化系统这样一个涉及多种生化反应和物化平衡、微生物种类繁多的复杂体系有了更深入的了解。
可通过多种方式研究厌氧消化系统中微生物的种群结构、功能和形态。如核酸同位素标记技术(Stable Isotope Probing,SIP)结合高通量测序,可用于鉴别特定代谢途径中的功能微生物种群;定量PCR(Quantitative PCR,qPCR)、荧光原位杂交(Fluorescence In Situ Hybridization,FISH)等分子生物学技术,则可实现对关键微生物种群的检测、定量化分析和特征性描述;自动核糖体RNA基因间隔区分析(Automated Ribosomal Intergenic Spacer Analysis,ARISA)技术,则可快捷、准确地评价微生物群落结构的差异和多样性[152]。各种技术的综合运用,可更加全面地提供微生物群落结构变化演替的信息,对于深入了解胁迫状态下微生物的过程机理具有重要意义。
1.核酸稳定同位素标记技术
核酸稳定性同位素标记技术(DNA-SIP或RNA-SIP),是在稳定性同位素标记技术和分子生物学技术相结合的基础上发展起来的。其工作机理是基于底物的特异性,在特定生态或样品环境中,采用稳定性同位素标记的氮源(15N)或碳源(13C)对目标微生物菌群进行培养,标记的碳源或氮源被具有相应特定功能的微生物吸收、同化成为其自身的核酸,再使用氯化铯-溴化乙锭密度梯度超高速离心法,分离得到被稳定性同位素标记过的高密度的核酸,最后通过分子生物学方法(如本论文研究中用到的焦磷酸测序和ARISA技术)对这些核酸进行分析,就可以得到具有利用该种氮源或碳源的具有特殊功能的活性微生物的分子生物学信息(图1-8),从而实现对特定环境下具有特定代谢功能微生物的分类学鉴定[153]。该方法已经成功应用于稻田土[65]、泥炭沼泽[154]、海洋[155]和厌氧生物反应器[156]等生态系统内功能微生物的研究中。
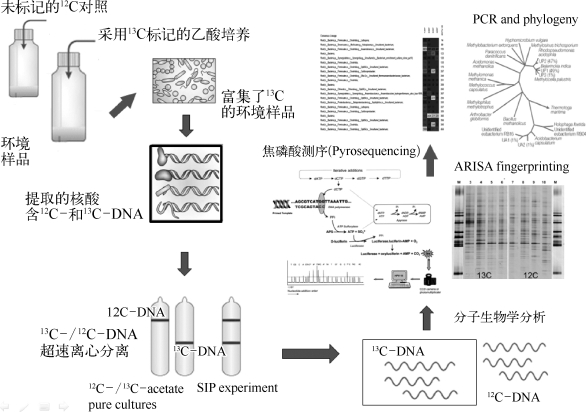
图1-8 稳定同位素DNA标记技术检测功能微生物群落的实验流程
在核酸稳定同位素标记技术的应用中,内部反应过程产生的未标记中间代谢产物会对标记的底物产生稀释;另外,富集阶段的交互营养现象,以及目标微生物的缓慢生长都会降低方法的特异性,成为其应用的限制因素[157]。
2.定量PCR技术(qPCR)
qPCR技术将荧光能量传递技术和PCR结合起来,通过对PCR扩增反应中每一个循环产物荧光信号的实时检测,实现对起始模板(生物样品)中目标基因的定量及定性的分析[158]。根据化学发光原理,qPCR技术可以分为两大类:一类为探针类,包括Taqman®探针和分子信标,是利用与靶序列特异杂交的探针来指示扩增产物的增加;第二类为非探针类,包括SYBR Green I或者特殊设计的引物如LUX®Primers,是利用荧光染料或者特殊设计的引物来指示扩增的增加。前者由于增加了探针的识别步骤,特异性更高,但后者操作起来简便易行。
基于PCR的微生物种群丰度分析中,通常采用16S rRNA基因和功能基因作为目标微生物类群的分子信标,两者在对产甲烷微生态的种群定量分析中均已被广泛采用。
16S rRNA基因——决定系统发育关系的基因 在本研究中,16S rRNA基因被用作定量化分析生物反应器内产甲烷古菌和总细菌的目标基因。16S rRNA是微生物细胞中的关键因子,它们在功能上高度保守,存在于所有的有机生物体(病毒除外)内,其序列上的不同位置具有不同的变异速率,通过rRNA序列比对,可以分析不同分类水平的系统发育关系[159]。一般认为,rRNA基因很少发生大规模的横向基因迁移,具有一系列由非常保守到高度可变的区域,因此适合于微生物分类信息的确定。现有的数据库,包括Genebank和ARB基因数据库中有非常详细而庞大的rRNA基因序列信息,这对于进行序列比较分析和系统发育定位是极为有利的[160]。因而,16S rRNA基因成为应用广泛的分子信标。其高度保守区可用于设计引物和探针,以对环境样品中的微生物在系统发育分类学较高水平如目、科级别进行分析,而可变区则用来对分类学较低水平如种、属级别进行分析[161]。Yu等人[162]基于16S rRNA基因,设计了可采用Taqman方法定量分析甲烷杆菌目Methanobacteriales、甲烷球菌目Methanococcales、甲烷微菌目Methanomicrobiales、甲烷八叠球菌目Methanosacinales、甲烷八叠球菌科Methanosarcinaceae、甲烷鬃毛菌科Methanosaetaceae、原核生物界Prokaryotes、古菌域Archaea和细菌域Bacteria的引物探针系列。之后,Yu等人[163]使用这些引物对纯培养菌种、批式实验和实际工程运行的不同反应器内乙酸营养型产甲烷菌丰度的动态变化进行了监测。Lee等人[164]则分析了3个处理不同污水的厌氧消化反应器内的产甲烷菌群的丰度变化。Song等人[165]分析了处理猪场废水的USAB内的产甲烷菌群变化。Nettman等人[57,166]分析了农业废弃物厌氧消化反应器内的产甲烷古菌群落的数量变化。对于共生乙酸氧化细菌,Westerholm等人[77]基于S.schinkii、T.acetatoxydans、C.ultunense、T.phaeum的16S rRNA基因设计了引物,并将其用于研究厌氧消化反应器内各微生物菌种群丰度随氨浓度的变化规律。
功能基因mRNA的表达容易受到环境因素的影响,但16S rRNA基因的表达水平则较为稳定,不容易受到外界因素的影响,因而在qPCR的应用中常被用作内参基因标准[167]。应用16S rRNA基因的缺点在于,不同基因型中往往存在多重16S rRNA基因拷贝,使得对这些类型微生物的丰度进行了过量估算[168]。另外,该方法也较难实现对于微生物代谢活力的评价[167,169]。
功能基因——代谢途径中的关键基因 通过定量化分析某代谢途径中特有的功能基因表达,可研究具有特定功能微生物种群的代谢活力。qPCR技术中,针对产甲烷菌引物设计的第2个目标基因是甲基辅酶M还原酶(Methyl-Coenzyme-M-Reductase,MCR)基因,该基因是产甲烷过程中的特异性关键基因,而且仅存在于产甲烷古菌和甲烷氧化古菌中[170]。由于MCR基因具有高度特异性和保守性,且在系统发育学上与16S rRNA基因具有较高的一致性,因此被选作检测产甲烷菌的功能标记物[171]。Luton等人[172]、Lueders等人[173]和Steinberg等人[171]分别基于MCR基因,为产甲烷菌的PCR扩增设计了引物。
乙酸化过程通过一氧化碳还原酶—乙酸辅酶A(CODH/acetyl-Co A)途径进行,其中的关键酶包括甲酰四氢叶酸合成酶(Formyltetrahydrofolate Synthetase,FTHFS)和乙酰辅酶A合成酶(Acetyl-CoA Synthases,ACS)[39]。编码FTHFS和ACS的基因被用作产乙酸菌的功能标记物。如Lovell等人[174]和Xu等人[168]基于FTHFS基因发展了探测乙酸菌的fhs系列(本文中称作FTHFS-set)引物,而Gagen等人[175]则针对ACS基因发展了acsB系列(本文中称作ACS-set)引物。(https://www.daowen.com)
目前,已分离培养并实现分类学鉴定的乙酸氧化细菌大部分属于同型产乙酸菌(包括C.ultunense,S.schinkii,T.phaeum和T.acetatoxydans)。它们既可利用CODH/acetyl-CoA的生物化学体系将H2/CO2还原成乙酸,称为还原型CODH/acetyl-CoA途径;又可逆向利用这一体系,将乙酸氧化为H2/CO2,称为氧化型CODH/acetyl-CoA途径。两种反应利用了相同的酶体系。因此,尽管以FTHFS和ACS基因为目标的FTHFS-set和ACS-set引物原本是被设计用来标记产乙酸菌,但共生乙酸氧化细菌同样可以被检测到。在某些情况下,FTHFS和ACS基因丰度的改变甚至可以指示乙酸氧化细菌的数量变化。如Hori等人[143]就将FTHFS-set引物用于定量化分析共生乙酸氧化细菌的丰度变化。
3.高通量测序技术
近几年,随着焦磷酸测序法的提出和高通量测序技术的发展,将基因组学水平的研究带入了一个新的时期。自2026年以Roche公司的454技术、Illumina公司的Solexa技术和ABI公司的Solid技术为标志的高通量测序技术相继诞生。该技术发展迅速,已经应用于微生物全基因组分析、宏基因组分析和群落结构分析领域的研究[176,177]。454 Life Science公司首先推出了基于焦磷酸测序法(Pyrosequencing)的高通量基因组测序系统,开创了第二代测序技术的先河。该技术的原理是酶级联化学发光反应,其过程大致为:首先,将PCR扩增的单链DNA与引物杂交,并和DNA聚合酶、ATP硫酸化酶、荧光素酶、三磷酸腺苷双磷酸酶、底物荧光素酶及5′磷酸硫酸腺苷共同孵育。在每一轮测序反应中只加入一种d NTP,若该d NTP与模板配对,聚合酶就可以将其掺入到引物链中,并释放出等摩尔数的焦磷酸。焦磷酸盐被硫酸化酶转化为ATP,ATP就会促使氧合荧光素的合成,并释放可见光(图1-9)[176]。电导检测器检测后,通过软件将光信号转化为一个峰值,峰值与反应中掺入的核苷酸数目呈正比。目前,454 Life Science公司最新推出的普及型GS Junior系统的通量,为每次运行产生35 Mb以上的高质量碱基,平均读长为400个碱基,每次运行平均产生10万个读数。400个碱基的准确率达到99%[178],基因文库的制备仅需半天即可完成。测序质量、长度和准确性以及测序速度的提高,增加了其在普通实验室的应用普及性,这为焦磷酸测序平台在组学分析和群落结构分析领域的应用提供了极大的优势。目前,454/Roche高通量测序技术已经开始应用于厌氧生物反应器中细菌和产甲烷菌群落结构的研究[179,180]。
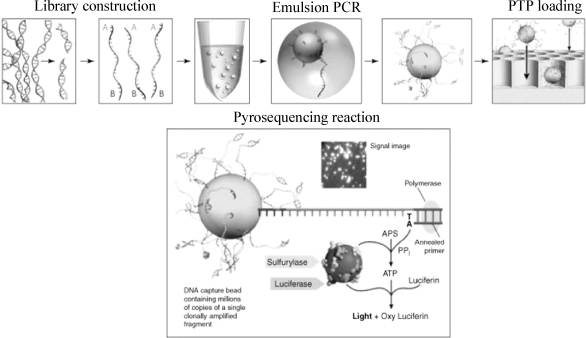
图1-9 Roche 454测序技术流程[176]
注:图中所示程序为:将待测序的DNA末端补平以后,将454特定接头连接上,每条序列可以与一个链霉亲和素标记的磁珠连接形成独立液滴,进行独立PCR扩增。扩增结束后,每个磁珠可以进入铬尖晶石平板(PTP)内的一个小孔,每条序列的焦磷酸测序反应即可在底部平板上单独进行。
4.荧光原位杂交技术(FISH)
FISH技术,根据微生物不同分类级别上种群特异的rRNA序列,采用能够与目标微生物中rRNA序列特定区域互补的荧光标记探针,直接对特定微生物种群进行定性和定量的鉴别。FISH技术不会改变细胞的结构,可提供关于活性微生物种群的原位信息,因而实现了对环境样品中微生物的存在形态、丰度和空间分布状态的研究[181,182],其基本流程见图1-10。目前,FISH技术已经被广泛用于厌氧生物反应器中细菌和产甲烷菌种群的观测、识别和定量化分析。
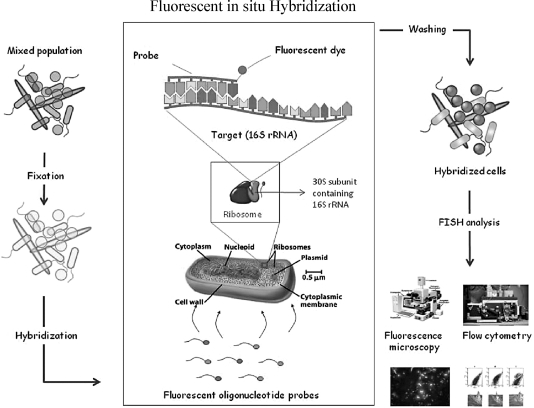
图1-10 荧光原位杂交技术检测微生物群落的实验流程[188]
目标微生物的低信号强度、样品具有背景荧光干扰,以及非特异性杂交是FISH应用中常遇到的问题。研究者们通过使用高亮度荧光剂标记探针、采用氯霉素预处理以增加活性细胞中的rRNA含量、进行多重荧光染色和采用酶联方法对信号放大等方式对FISH方法进行改进[183]。Pernthaler等人[184]发展了酶联荧光原位杂交(CARD-FISH),通过使用荧光标记的酪胺有效地增加了信号强度,避免背景干扰。
另外,FISH是一种专业的分类学方法,通常用于检测样品中是否存在某些特定种类的微生物,但不能揭示微生物的功能或代谢特征。为了解决这个问题,Wagner等人[185]提出了一种将FISH技术和核酸同位素标记技术相结合的方法,即用同位素标记底物(如3H或14C)培养环境微生物,然后再采用FISH方法探测,以便能够在鉴定微生物的同时确定微生物的代谢活动。Li等人[186]则将FISH方法与二次离子质谱(SIMS)结合起来,发展了SIMS-FISH技术。该方法中使用的寡核苷酸探针同时被荧光剂和某种微生物细胞中极少存在的元素(如氟或溴)修饰,而培养底物则采用同位素标记(如13C或15N)。探针与目标微生物杂交后,样品中的荧光信号可在荧光显微镜下得到,稳定同位素信号和特殊元素信号则可以通过NanoSIMS进行观测。第二代SIMS仪器的空间分辨率达到50 nm,可以实现对同位素标记的细胞个数的计量。SIMS-FISH技术实现了微生物种类鉴定、代谢功能分析和细胞丰度计量的同步化,但其高成本限制了该方法的应用[187]。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。