实验目的
2.学习并掌握基因组织定位方法。
实验原理
绿色荧光蛋白(Green fluorescent protein,GFP)是20世纪60年代在水母中发现的发光蛋白,是由238个氨基酸组成的单链多肽,在蓝光或紫外光激发下发出绿色荧光,用于蛋白质在活细胞中的准确定位及动态变化观察。将目的基因与载体上带有的GFP基因融合,转化拟南芥后,融合蛋白可以在植物体内被表达,通过荧光共聚焦显微镜可以直接检测得到目的基因表达产物在细胞中的定位情况。
GUS(b-glucuronidase)基因作为一种报告基因,在植物遗传转化研究中有广泛的用途。GUS基因来自于大肠杆菌,可以编码b-葡聚糖苷酶(一种水解酶),能催化底物5-溴-4-氯-3-吲哚葡聚糖醛酸苷(5-bromo-4-chloro-3-indolyl-glucronide,X-Gluc)分解产生肉眼可见的深蓝色化合物,将目的基因特异性启动子与载体上带有的GUS 基因融合,转化拟南芥后,可以观察转基因植物中外源目的基因在植物组织器官中的表达情况。
实验用品
1.材料
GFP转基因拟南芥种子、GUS转基因拟南芥种子和野生型种子。
2.试剂
(1)1/2MS固体培养基
(2)10%NaClO
(3)无菌水
(4)90%丙酮
(5)GUS染色所用溶液
母液:0.2mol/LNaH2PO4,0.2mol/LNa2HPO4,250mmol/LK3Fe(CN)6,250mmol/LK4Fe(CN)6。其中NaH2PO4和Na2HPO4高压灭菌,K3Fe(CN)6和K4Fe(CN)6过滤除菌。
漂洗液:50mmol/LNa3PO4,2.5mmol/LK3Fe(CN)6,2.5mmol/LK4Fe(CN)6,0.1%Triton X-100,pH7.0。
染色液:漂洗液加2.0mmol/LX-Gluc。
3.器具
荧光共聚焦显微镜、解剖镜、恒温水浴锅、真空浓缩仪、显微拍照系统、电子天平、培养皿、1.5mL离心管等。
实验操作程序
(一)GFP荧光观察
1.GFP转基因拟南芥种子和野生型种子在1/2MS固体培养基上竖直培养无菌苗。
2.培养至第5d,在荧光共聚焦显微镜下直接观察根的伸长区(激发光波长488 nm)。
(二)GUS染色观察
1.将待染色的组织(如叶片、花序、角果、幼苗等)置于1.5mL离心管中,加入90%丙酮,使不同植物组织完全浸没,固定5~10min。
2.移去丙酮溶液,加入漂洗液漂洗2次。
3.移去漂洗液,加入染色液浸没植物组织。
4.抽真空3次,每次5min,移去所有气泡。
5.37℃孵育3h至过夜。
6.看见有明显蓝色可停止反应,移去染色液,加入ddH2O漂洗3~4次,在解剖显微镜下观察,拍照。
预期实验结果(www.daowen.com)
分别通过荧光共聚焦显微镜和体式解剖镜检测到GFP和GUS转基因拟南芥中,外源目的基因在拟南芥组织器官(侧根)中的表达情况如图19-Ⅴ-1所示。
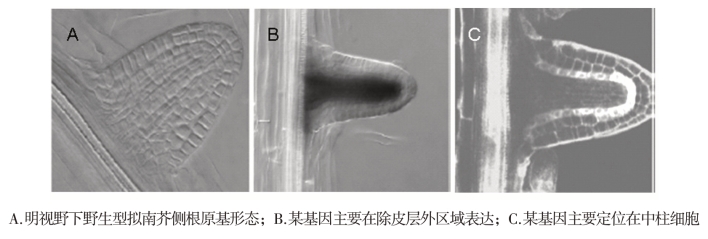
图19-Ⅴ-1 GFP荧光与GUS染色
(引自Hirota et al.,2007)
要点及注意事项
1.观察拟南芥中稳定表达的GFP融合蛋白常选用幼苗根部或下胚轴区域,这样可以减少叶绿体自发荧光的干扰。
2.GUS染液有毒,使用中需要注意安全。
3.GUS染色时间不宜过长,染色太深会影响结果。
4.GUS染色实验需要对植株各个器官都进行染色,包括幼苗、莲座叶、茎生叶、花和角果等。
作业与思考题
1.GFP激发光波长是多少?
2.GUS染色实验中丙酮的作用是什么?
1.金美芳.拟南芥总RNA的简便提取与效果分析[J].福建师范大学福清分校学报,2007,2:16-18.
2.李志邈,张海扩,曹家树,等.拟南芥激活标记突变体库的构建及突变体基因的克隆[J].植物生理与分子生物学学报,2005,31(5):499-506.
3.刘金亮.拟南芥实验室的常用种植方法[D].兰州:西北师范大学,2013.
4.阮燕晔,张莹,王波.拟南芥atcwinv1基因T-DNA插入纯合突变体PCR鉴定及表型观察[J].河南农业科学,2011,40(5):62-66.
5.张小红,许鹏博,郭萌萌,等.拟南芥G蛋白α亚基GPA1互作蛋白铜离子结合蛋白AtBCB的鉴定及功能分析[J].作物学报,2013,21(1):91-118.
6.赵淑清.一种筛选拟南芥突变体的有效方法[J].遗传,2001,23(3):260-262.
7.郑岑,张立平,唐忠辉,等.TAIL-PCR技术及其在植物基因中的克隆[J].基因组学与应用生物学,2009,28(3):544-548.
8.Hirota A , Kato T , Fukaki H, et al.The auxin-regulated AP2/EREBPgene PUCHI is required for morphogenesis in the early lateral root primordium of Arabidopsis[J].Plant Cell,2007,19:2156-2168.
9.Liu J , Zhu J K.A calcium sensorhomolog required for plant salt tolerance [J].Science,1998,280:1943-1945.
10.Liu Y G, Chen Y, Zhang Q.Amplification ofgenomic sequences flanking T-DNAinsertions by thermal asymmetric interlaced polymerase chain reaction[J].Methods Mol Biol,2005,286:341-348.
11.Meinke D W,Cherry J M,Dean C,et al.Arabidopsis thaliana:a model plant forgenome analysis[J].Science,1998,282:662,679-682.
12.Meins F Jr,Si-Ammour A,Blevins T.RNAsilencing systems and their relevance to plantdevelopment[J].Annu Rev Cell Dev Biol,2005,21:297-318.
13.Osato Y, Yokoyama R, Nishitani K.A principal role for AtXTH18 in Arabidopsis thaliana rootgrowth: a functional analysis using RNAi plants[J].J Plant Res,2006,119:153-162.
14.Poulsen L R, López-Marqués R L, McDowell S C, et al.The Arabidopsis P4-ATPase ALA3 localizes to the Golgi and requires a beta-subunit to function in lipid translocation and secretory vesicle formation[J].Plant Cell,2008.20:658-676.
15.Shindo C,Bernasconi G, Hardtke C S.Naturalgenetic variation in Arabidopsis: tools,traits and prospects for evolutionary ecology[J].Ann Bot,2007,49:1043-1054.
16.Tang C, Toomajian C, Sherman-Broyles S, et al.The evolution of selfing in Arabidopsis thaliana[J].Science,2007.317:1070-1072.
17.Yue S,Zhang W, Li F L, et al.Identification andgenetic mapping of four novelgenes that regulate leafdevelopment in Arabidopsis[J].Cell Res,2000,10:325-335.
(牛月)
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。







