
实验目的
1.学习并掌握拟南芥T-DNA插入突变体筛选目的和过程。
2.掌握TAIL-PCR鉴定T-DNA插入序列信息的原理和方法。
实验原理
拟南芥T-DNA插入突变体高通量(全基因组)筛选方法多样,需根据筛选目的而定。目前筛选突变体几乎都需要利用生理生化等方法定向鉴定表型获得。如利用根弯曲筛选环境胁迫相关的突变体,就是利用根的向地性生长会受到培养基中胁迫因子(如高盐、重金属离子等)的影响而停止生长的原理,可以筛选到相关的抗性和敏感突变体。将通过表型初筛得到的F0代种植后单株收种,得到的F1代就可以鉴定目的基因了。
CTAB(十六烷基三甲基溴化铵,hexadecyltrimethylammonium bromide)是一种阳离子去污剂,具有从低离子强度溶液中沉淀核酸与酸性多聚糖的特性。在高离子强度的溶液中(>0.7mol/LNaCl),CTAB 与蛋白质和多聚糖形成复合物,但不能沉淀核酸。通过有机溶剂抽提,去除蛋白、多糖、酚类等杂质后,加入乙醇沉淀即可使核酸分离出来。通常采用机械破碎植物细胞,然后加入CTAB分离缓冲液将DNA溶解出来,再经氯仿-异戊醇抽提除去蛋白质,最后得到DNA。
TAIL-PCR(thermal asymmetric interlaced PCR)是一种分离与已知序列邻近的未知DNA序列的分子生物学技术,可以用来鉴定植物基因组中T-DNA的插入位点。该技术利用3个根据已知Ti载体序列设计的嵌套特异性边界引物(TR或TL)分别和简并引物AD组合进行PCR反应,选择恰当退火温度对目标片段进行PCR扩增。TAIL-PCR共分3次反应。在首轮PCR中,以AD/TR1为引物扩增右边界(以AD/TL1扩增左边界),然后取首轮PCR产物为模板,以AD/TR2(AD/TL2)进行二次PCR,再以二次PCR产物为模板,AD/TR3(AD/TL3)为模板进行第三轮PCR,将第2、3轮的PCR产物进行电泳分析,可以从突变体中获得带有T-DNA左右边界的旁侧序列,再通过序列比对得到T-DNA插入序列信息。
实验用品
1.材料
野生型拟南芥种子、拟南芥T-DNA插入诱变突变体库种子。
2.试剂
(1)2×CTAB提取缓冲液:100mmol/LTris-HCl(pH8.0),20mmol/LEDTA,1.4mol/LNaCl,2%(W/V)CTAB,40mmol/L巯基乙醇,灭菌。
(2)异丙醇
(3)10×PCR buffer
(4)25mmol/LMgCl2
(5)dNTPs mixture
(6)Taq 酶
(7)氯仿-异戊醇(24:1)
(8)70%乙醇
(9)液氮
(10)引物:
pBIB-BASTA TR1: CCA TCG CCC TGA TAG ACG GTT T
pBIB-BASTA TR2: CAA CCC TAT CTC GGG CTA TTC TT
pBIB-BASTA TR3: CCG CTT GCT GCA ACT CTC TCA
AD1 Primer: NTC GAS TWT SGW GTT
AD2 Primer: NGT CGA SWG ANAWGA A
AD3 Primer: WGT GNAGWA NCA NAG A
实验操作程序
(一)拟南芥T-DNA插入突变体筛选
在用除草剂BASTA 筛选转化植株F0代时,会出现一些直接可观察到表型的突变体植株,拍照、单株收种,待后续插入基因鉴定。但绝大多数F0代植株不表现特殊表型,需要用一些筛选方法定向选择突变体,如抗逆性筛选等。将通过表型初筛得到的F1代种植后单株收种,得到的F2代就可以进行插入基因鉴定。
(二)提取基因组DNA
1.取1~50g新鲜植物材料(如嫩叶),于液氮中快速研成粉末。
2.将粉末转入预冷的1.5mL离心管中,立即加入等体积2%CTAB提取缓冲液,65℃保温1h,其间不时摇动。(www.daowen.com)
3.加入等体积的氯仿-异戊醇(24∶1),轻缓颠倒混匀,12 000r/min离心10min。
4.将上清液转入另一离心管中,加入等体积的氯仿/异戊醇,颠倒混匀,12 000r/min离心5min。
5.将上层水相转入新的离心管中,加入0.7体积的异丙醇,混匀,室温下放置30min。
6.12 000r/min离心10min,弃去上清液。
7.70%乙醇漂洗2次,12 000r/min离心2min,沉淀吹干。
8.风干后加入30~50μL的TE缓冲液溶解DNA,-20℃保存备用。
(三)TAIL-PCR
1.3次PCR反应分别按表19-Ⅲ-1加样体系进行。
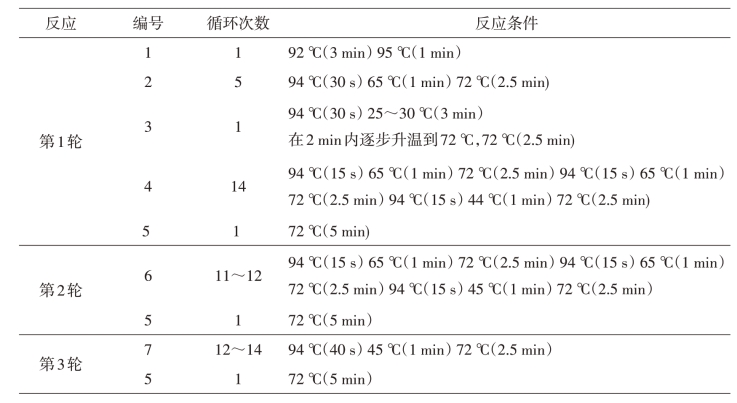
2.按表19-Ⅲ-2设定PCR反应参数。
3.将第2轮和第3轮反应产物分别在浓度1.0%的琼脂糖凝胶上样,进行凝胶电泳(参照实验十八-Ⅲ中PCR检测方法)。当第2轮比第3轮反应产物略大,约为TR2和TR3引物间区段大小时,可以扩大反应体系至50μL,重新扩增。
表19-Ⅲ-1 PCR反应加样体系
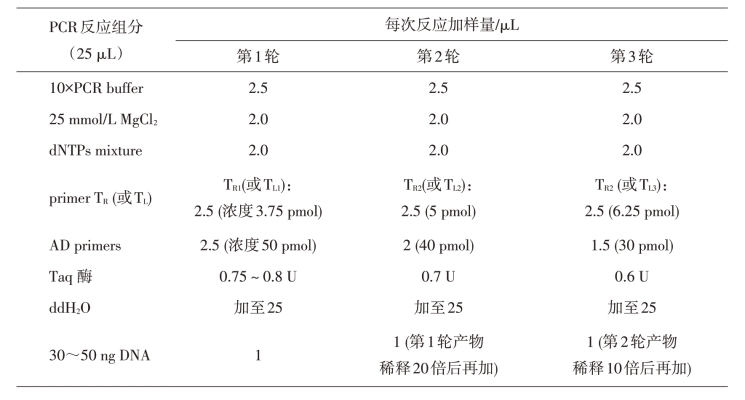
表19-Ⅲ-2 PCR反应参数
4.第3轮反应产物通过凝胶电泳分离后,将条带切割下来放入干净的1.5mL离心管中,直接送去生物公司测序或者进行凝胶回收后再送样测序。
5.测序后得到的序列信息可以直接在网站( http://www.arabidopsis.org/)上通过Tools>BLAST进行序列比对,最终得到详细的插入基因信息。
预期实验结果与分析
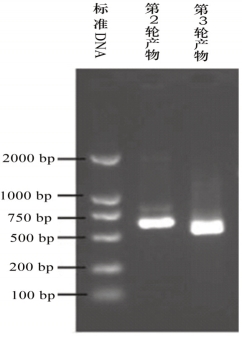
如果扩增到目的片段,就会在电泳中同时出现第2、3轮反应的条带(图19-Ⅲ-1),且第2轮产物条带较靠近点样孔。
要点及注意事项
1.突变体表型筛选时必须同时有野生型和未处理组作为对照实验。
2.提取DNA的所有操作均需温和,避免剧烈震荡。
3.所用有机溶剂均对人体有害,需要在通风橱中进行。
4.抽提上层水相时要小心,切勿吸取中间白色带(主要是蛋白和核酸)造成DNA污染。
5.提取DNA沉淀要尽量干燥,完全去除有机溶剂(主要是乙醇)。
作业与思考题
1.提取DNA过程中加有机溶剂的作用是什么?
2.TAIL-PCR反应后如何选择有意义的实验结果?

图19-Ⅲ-1 TAIL-PCR第2、3轮产物电泳
(引自Liu et al.,2005)
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。





